Projet DynaMo coordonné par Violette Thermes (INRA - LPGP)
Financement ANR JCJC, 2018 - 2021.
La maîtrise de la quantité d’œufs produits à chaque cycle reproducteur (fécondité) est un enjeu important que ce soit pour la gestion des populations sauvages de poissons ou en tant que levier d’amélioration de la compétitivité des élevages aquacoles. La dynamique globale de l’ovogenèse (taux et fréquence de recrutement, puis de croissance des ovocytes au sein de l’ovaire) peut être extrêmement variable que ce soit entre les différentes espèces de poissons, qui présentent des fécondités très variables, mais également au sein d’une même espèce en fonction des conditions environnementales. Les mécanismes qui gouvernent la dynamique de recrutement/croissance des ovocytes durant l’ovogenèse restent toutefois à ce jour peu connus, essentiellement en raison de difficultés méthodologiques inhérente à l’histologie classique (2D sur coupes) qui ne permet pas d’accéder facilement à la totalité des ovocytes à l’échelle de l’ovaire entier.
Chez le médaka, un petit poisson d’aquarium à cycle de reproduction court utilisé pour étudier l’ovogenèse, nous avons récemment mis en évidence une vingtaine de miARNs exprimés de façon prédominante dans l’ovaire, dont un miARN (miR-202) jouant un rôle crucial pour le succès reproducteur femelle et notamment la fécondité. Les miARNs sont des régulateurs fins de l’expression des gènes qui sont impliqués dans le contrôle de nombreux processus biologiques et les données préliminaires dont nous disposons indiquent qu’il jouent un rôle important dans la régulation de la fécondité chez le médaka.
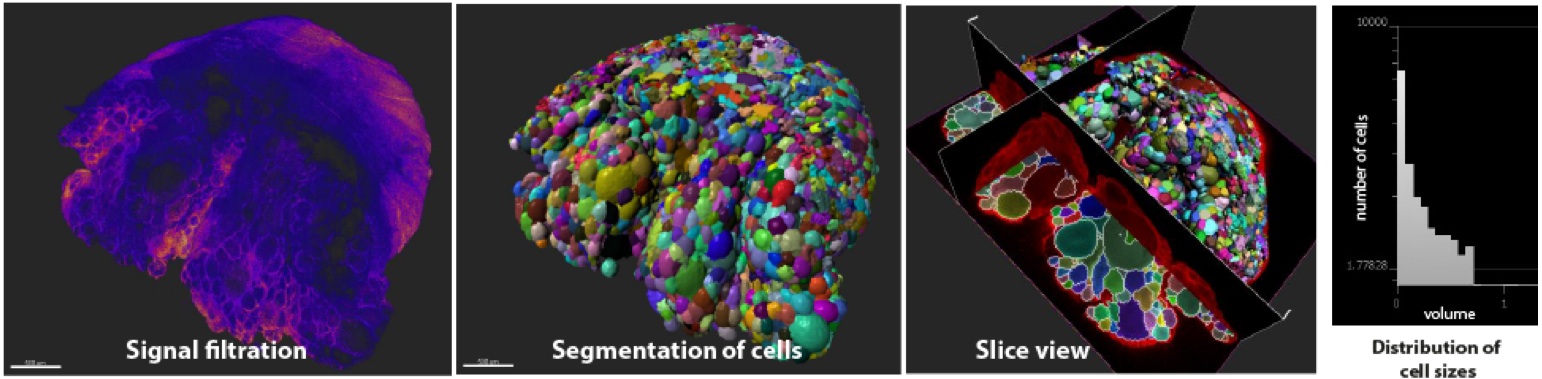
L’objectif du projet DynaMo est de comprendre précisément non seulement la dynamique globale de l’ovogenèse mais aussi le rôle des miARNs dans ce processus. Notre ambition est ici d’aller au delà d’une simple liste de miRNAs régulateurs de l’ovogenèse en fournissant une vision précise de ce processus intégrant la dynamique cellulaire et ses régulations par les miRNAs. Dans ce but, nous associons une approche descriptive (par imagerie 3D), une approche fonctionnelle (grâce aux techniques d’édition du génome) et une approche de modélisation mathématique.



